原文發表於第 11 屆 iT 邦幫忙鐵人賽 (https://ithelp.ithome.com.tw/articles/10219913)
自動修剪機,修鹼基
fastq 檔案中存放的資料包括序列及其對應的品質,雙端定序的結果分別存放在結尾標記 -1 和 -2 的檔案中。必須將定序品質不佳的鹼基剔除掉,才可以進行後續的分析。
Trimmomatic 是一款 java 撰寫的跨平台軟體,透過終端機指令提供參數來使用。

點選最新版本的 binary 即可,該頁面下方就有快速上手教學
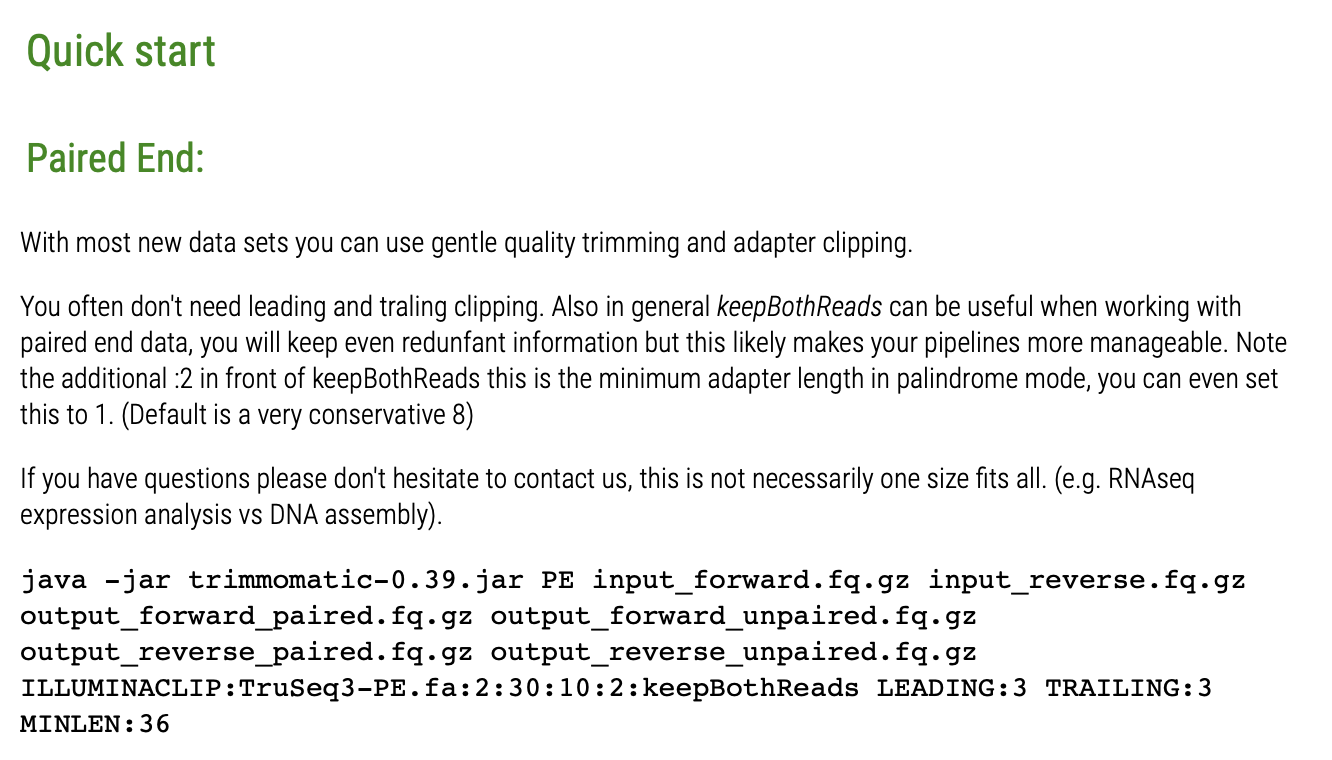
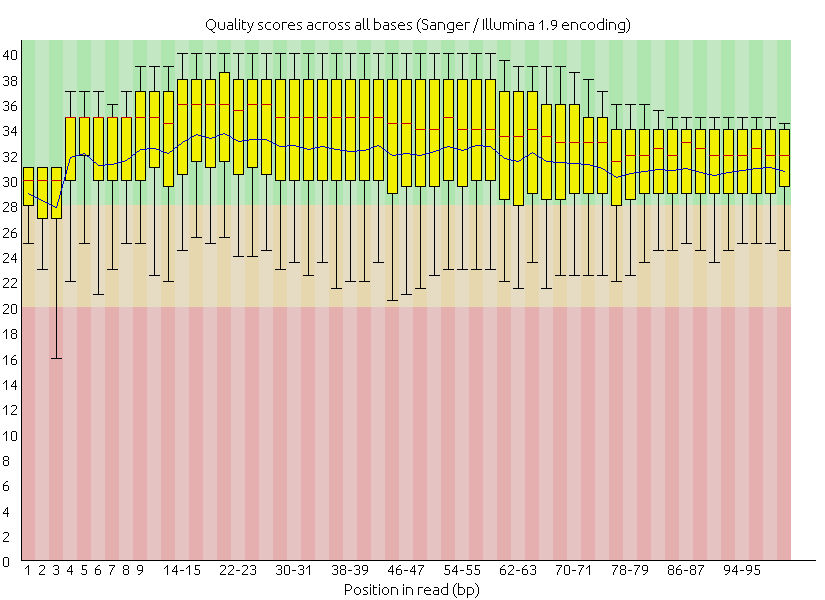
先來一段簡單的示範,下一段落才有實際手把手操作。原本品質差的序列以 FastQC 視覺化品質之盒方圖,可見許多位於末端的鹼基之定序品質已經來到紅色的不良範圍
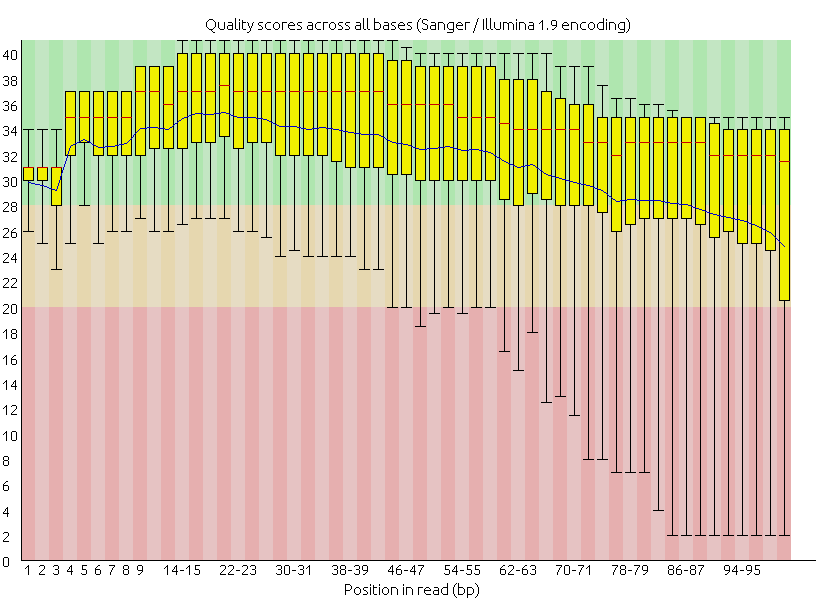
我們修剪的是雙端定序檔案,同一對序列來自同一條 DNA 的兩端,中間間隔 100 至 300 不等的未知序列。因此每一對序列的修剪過程與產出檔案如下:
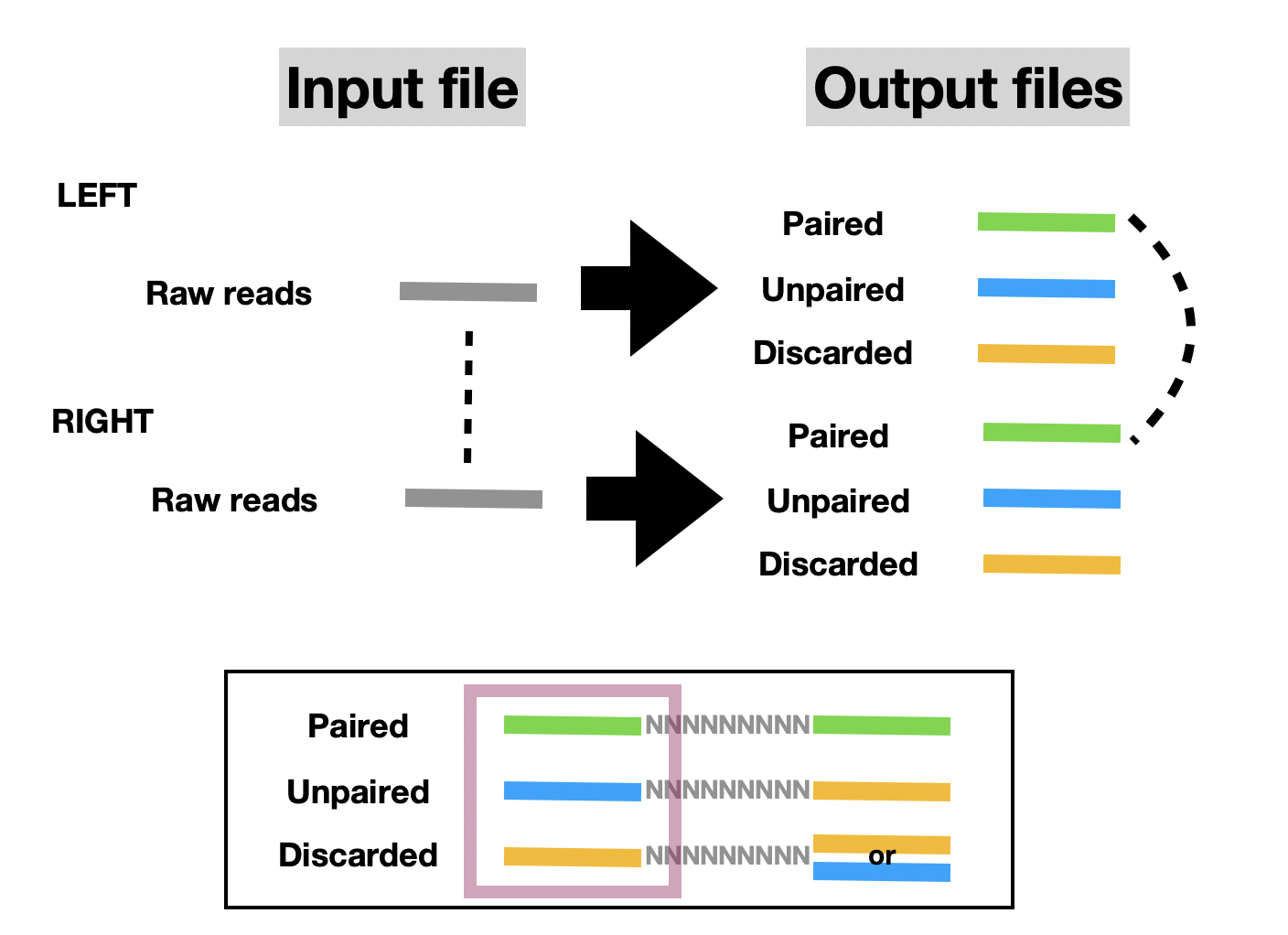
也就是說,輸入一對 (兩組) 序列檔案,會跑出一對+兩組 (共四組) 序列檔案,一對成對與兩組非成對。
- 成對,可喜可賀,兩端品質都過關,修成正果。
- 非成對,一方有情,一方無意,留下來的人最傷心。
- Discarded,無明眾生難逃苦難,已經投入輪迴不再露面。
在修剪後,不論是成對或非成對的修剪結果,品質都來到了安全的綠色區塊
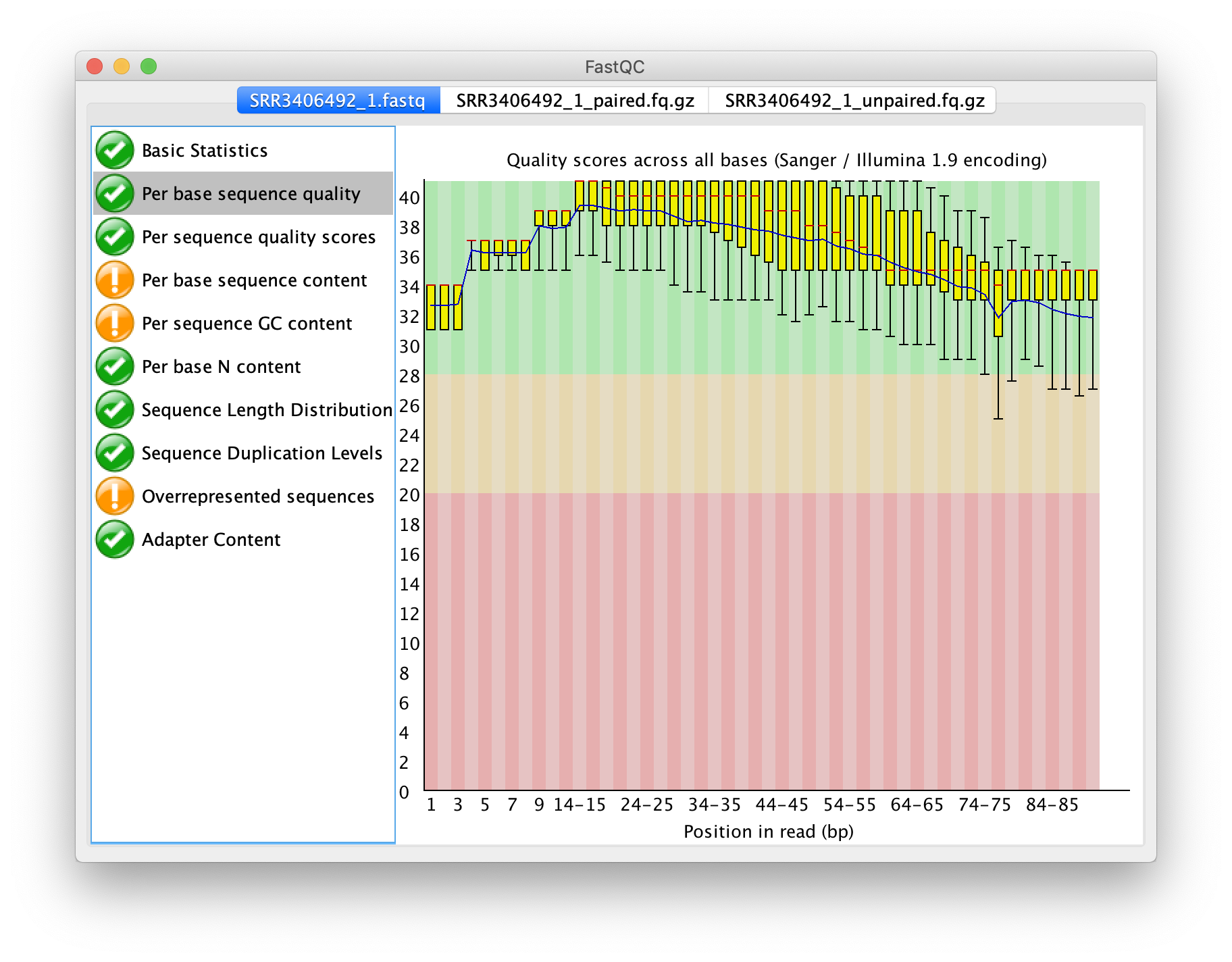
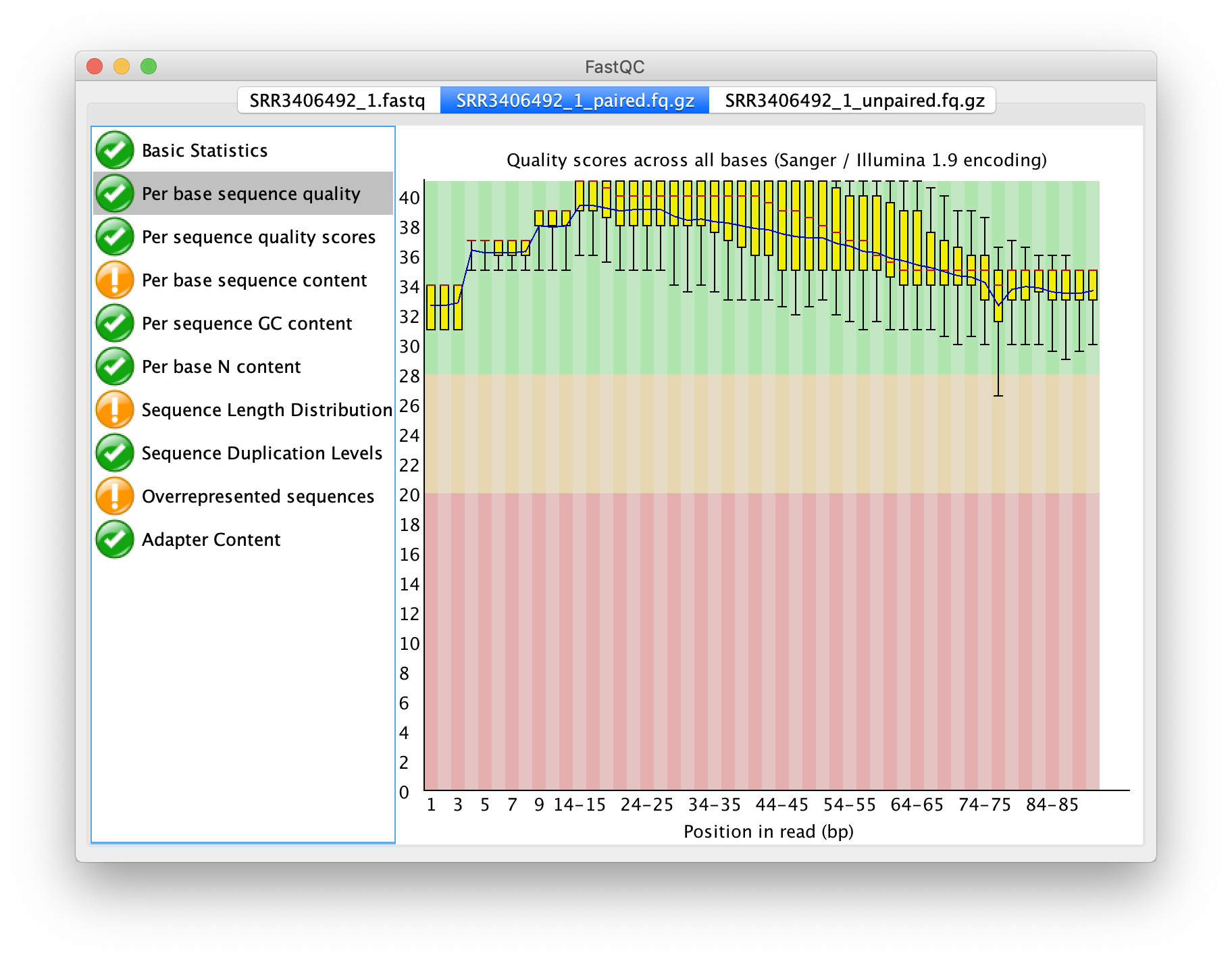
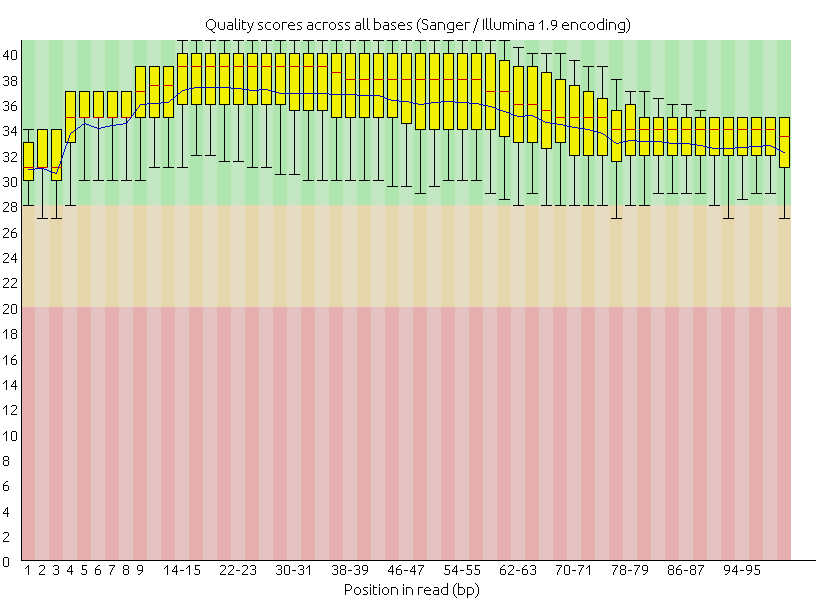 輸出之成對序列品質之盒方圖
輸出之成對序列品質之盒方圖
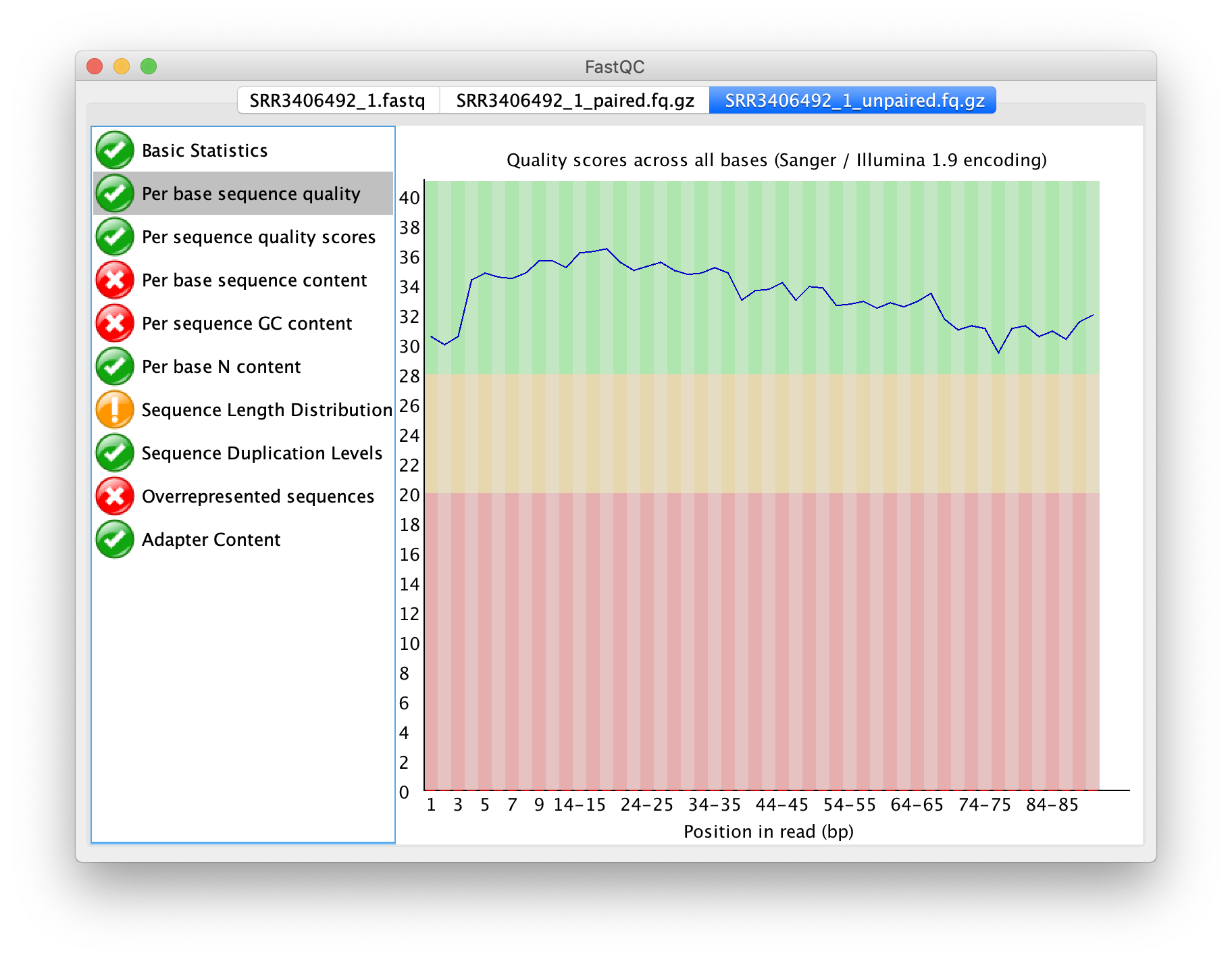
 輸出之非成對序列品質之盒方圖
輸出之非成對序列品質之盒方圖
實戰演練示範
手癢想試試看吧?一樣以線上資料練習,以下方指令下載,僅有五千條讀序 (spot)
fastq-dump -X 5000 --split-files SRR3406492
這個練習中以 SRA 上的資料示範,但是這是已經被修剪過的檔案,因此品質極佳,輸出結果僅供參考。
以下列的預設指令修剪,每個參數所代表的意思,以淺綠底色標注在其旁邊

指令複製區!在 trimmomatic-0.39.jar 之目錄中執行,要修剪的原始檔案放在其上一層,記得注意軟體版本名稱、輸入輸出檔案之名稱要自行更改:
java -jar trimmomatic-0.39.jar PE -phred33 ./../SRR3406492_1.fastq ./../SRR3406492_2.fastq SRR3406492_1_paired.fq.gz SRR3406492_1_unpaired.fq.gz SRR3406492_2_paired.fq.gz SRR3406492_2_unpaired.fq.gz ILLUMINACLIP:./adapters/TruSeq3-PE.fa:2:30:10 LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 MINLEN:36
共輸出四個檔案,操作者要自行命名。執行成功會得到如下的回傳結果,五千條序列中有 98.84% 都被留下來了!
(base) Benjamins-Device:Trimmomatic-0.39 bendjamin101001$ java -jar trimmomatic-0.39.jar PE -phred33 ./../SRR3406492_1.fastq ./../SRR3406492_2.fastq SRR3406492_1_paired.fq.gz SRR3406492_1_unpaired.fq.gz SRR3406492_2_paired.fq.gz SRR3406492_2_unpaired.fq.gz ILLUMINACLIP:./adapters/TruSeq3-PE.fa:2:30:10 LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 MINLEN:36
TrimmomaticPE: Started with arguments:
-phred33 ./../SRR3406492_1.fastq ./../SRR3406492_2.fastq SRR3406492_1_paired.fq.gz SRR3406492_1_unpaired.fq.gz SRR3406492_2_paired.fq.gz SRR3406492_2_unpaired.fq.gz ILLUMINACLIP:./adapters/TruSeq3-PE.fa:2:30:10 LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 MINLEN:36
Multiple cores found: Using 4 threads
Using PrefixPair: 'TACACTCTTTCCCTACACGACGCTCTTCCGATCT' and 'GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT'
ILLUMINACLIP: Using 1 prefix pairs, 0 forward/reverse sequences, 0 forward only sequences, 0 reverse only sequences
Input Read Pairs: 5000 Both Surviving: 4942 (98.84%) Forward Only Surviving: 45 (0.90%) Reverse Only Surviving: 11 (0.22%) Dropped: 2 (0.04%)
TrimmomaticPE: Completed successfully
再次以 FastQC 檢測序列品質,成對之輸出,僅比原本的狀況提升一點點
非成對之輸出
以上就是 Trimmomatic 的使用紀錄,如果有任何安裝與使用上的問題,需要中文支援,歡迎在下方留言~
參考資料與延伸閱讀
USADELLAB.org - Trimmomatic: A flexible read trimming tool for Illumina NGS data
